如何使用gift比较两组不同被试在不同扫描条件下的DMN差异?
Submitted by qizhi on Fri, 09/23/2016 - 21:10
你好,我目前正在研究两组数据间的差异。打算比较1.5T和3.0T不同机器扫描的数据间差异,新手一枚,望多指教!!!
存在几个问题,也顺便想问问楼主。
1、1.5T机器扫描的被试有10个,3.0T扫描的被试是另外的10subject。若想比较DMN的差异,是否有意义?
2、使用gift软件若要进行组独立成分分析group ICA,如何对这两组不同被试的数据进行group ICA呢? 目前,我只知道若是同样的10个subject的话,可以在数据输入的时候 选择2 session of 10subject。最后生成所有数据的mean component, 那要是1.5T和3.0T扫描的10个被试不同的要怎么进行group ICA呢?又如何进行DMN比较呢?一般是比较采用什么统计方法对何种差异进行比较?
Forums:
- Read more about 如何使用gift比较两组不同被试在不同扫描条件下的DMN差异?
- 3 comments
- Log in or register to post comments
- 7430 reads

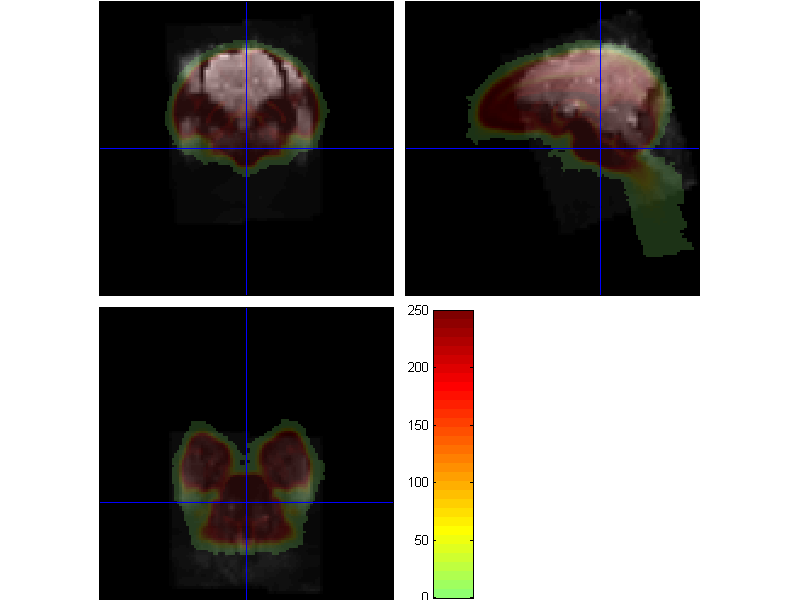 ,配准查看结果如下。如何提取脑组织啊,本人尝试用了fsl软件中的BET工具提取脑组织,但是结果不太理想,似乎去除了脑周围的部分,但是脖颈仍然未去除。请问,老师们怎么成功提取脑组织部分呢?
,配准查看结果如下。如何提取脑组织啊,本人尝试用了fsl软件中的BET工具提取脑组织,但是结果不太理想,似乎去除了脑周围的部分,但是脖颈仍然未去除。请问,老师们怎么成功提取脑组织部分呢?