Submitted by marvelancet on Sat, 10/02/2010 - 23:17
亲爱滴们:
今天碰到这样一个问题,焦头烂额:
由于这批数据需要手动配准,所以我的处理流程如下:
1. 用dparsf批处理至realign。
2.用spm做coregister.
3.用AFNI转换格式后做Normalize
4.转换个格式后,将得到的EPI相文件放入FunImgNormalized文件夹。
然后启动dparsf,
先跳过Smooth做的ReHo,很顺利,detrend, filter, reho...没有任何问题。
然后继续用dparsf做alff以及falff,操作过程如下:
-matlab下输入dparsf
-依次去掉smooth前所有的勾
-选择工作目录
- 输入时间点190
- 选择自定义模板

- 按run
然后提示我文件夹下首个被试的time point为0,
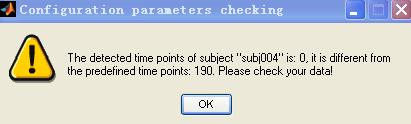
若将time point改为0,则提示需要定义time point,
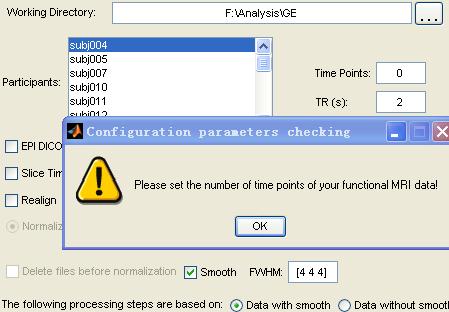
总之就是做不下去。
尝试将FunImgNormalized目录下,被试文件夹中EPI像文件prefix改为"w",
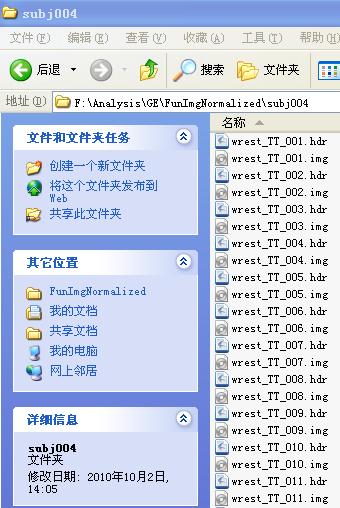
同样的提示,不能执行。
只要选中smooth就提示0,做不下去。
而跳过smooth则后面的detrend等步骤不受影响。
我在windows和linux环境下都做过了,一样的结果。
怎么回事呢?有没有碰到类似情况的坛友?怎样解决?
请教了几个,都说没见过。。。
是不是一个潜在的bug?
marvel

Submitted by marvelancet on Sun, 10/03/2010 - 21:41 Permalink
明白了
多谢超赣提醒
做smooth前,确认FunImgNormalized目录中被试源文件开头为“wra*”
我用AFNI后用MricroN转格式,会生成前缀“f*”所以出现这个问题。
用dparsf中utility改名就好了。
marvel
Submitted by YAN Chao-Gan on Mon, 10/04/2010 - 13:20 Permalink
Re
Thanks.
考虑在下一版中在此处取消wra*的严格要求。