关于DPARSF处理数据中间环节出错
老师们好,我有几个问题想请教大家。
1、在DPARSF跑数据中,detrend这一步出错,Warning: Invalid escape sequence appears in format string. See help sprintf for valid escape sequences.
- Read more about 关于DPARSF处理数据中间环节出错
- Log in or register to post comments
- 3970 reads
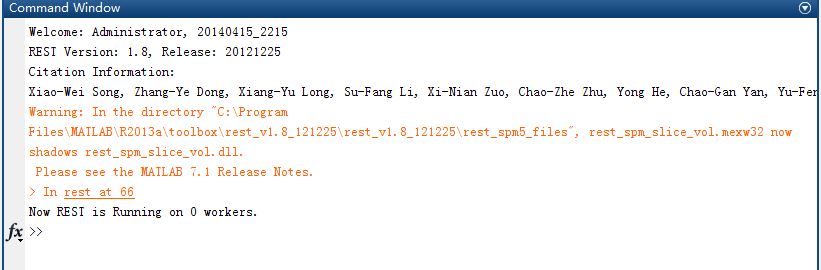
启动rest时出现警告
请教老师,我启动rest时总是出现下面警告,是怎么回事?
- Read more about 启动rest时出现警告
- Log in or register to post comments
- 3603 reads
DPARSFA 2.3 - new segment + DARTEL
Dear all,
I run DPARSFA starting with nifti files and so far it went more or less okay, but I get error messages for the new segment + DARTEL step:
Error using cfg_util (line 835)
Job execution failed. The full log of this run can be found in MATLAB command window, starting with
- Read more about DPARSFA 2.3 - new segment + DARTEL
- 2 comments
- Log in or register to post comments
- 9297 reads
Error message: "Too many .nii.gz files in each subject's directory, should keep one 4D .nii.gz file."
Hi, I am trying to set up DPARSFA to begin preprocessing my data. But when I click "Run," an error message tells me:
"Too many .nii.gz files in each subject's directory, should keep one 4D .nii.gz file."
DynamicBC_V1.0
- Read more about DynamicBC_V1.0
- 1 comment
- Log in or register to post comments
- 8982 reads
DPARSF後進行GIFT的問題
在SPM8可以成功進行到GROUP ICA 20個成分的結果
使用DPARSF過濾CSF得到的NIFTI檔 在跑GIFT的時候 14個人無法同時進行GIFT分析
會分成特定10個一起可以跑 另4個一起跑也可以 但都只能跑10個成分的結果 讓14個合併就不行
不確定是甚麼原因
錯誤訊息為
Error using load
Unable to read file 'D:\TEST_pca_r2-1.mat': no such file or directory.
而且Display幾乎沒有紅亮點 這樣是甚麼意思呢?
試了好多天 希望能為我解答 感激不盡
- Read more about DPARSF後進行GIFT的問題
- 2 comments
- Log in or register to post comments
- 6503 reads
rest 单样本t检验出现负值求助
各位老师好!
我在用rest对一组mALFF做单样本t检验得到mALFF Tmap, 然后用slive viewer进行查看 得到的图像里面有负值,采用的FDR双尾矫正,Q值为0.001,单样本T检验出现负值
查看显著脑区不是应该只有正值吗 怎么会出现负值呢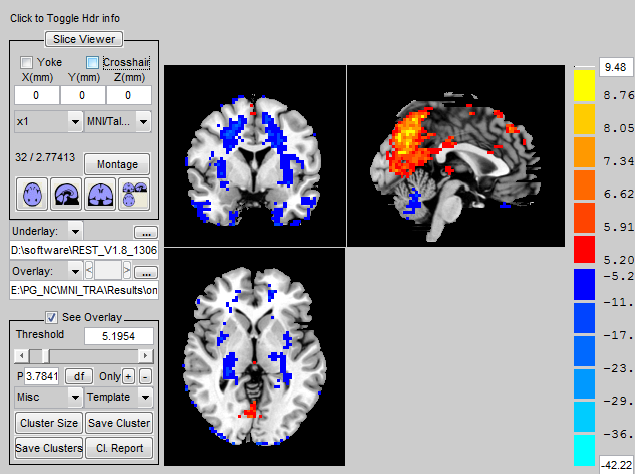
- Read more about rest 单样本t检验出现负值求助
- 5 comments
- Log in or register to post comments
- 14472 reads
北师大多模态神经影像与脑连接组学专题培训班开始报名了
大家好,
- Read more about 北师大多模态神经影像与脑连接组学专题培训班开始报名了
- Log in or register to post comments
- 4002 reads
DPARSFA 报错
出错内容如下 win2008,Matlab2011b ,DPARSF V2.2_121225,Rest 1.8
任务设置文件见附件
多谢指教!
Error using y_ExtractROISignal (line 188)
Wrong ROI definition, please check:
D:\FTP\Process_new\Masks\20121121002_BrainMask_05_91x109x91.nii
Error in DPARSFA_run>(parfor body) (line 1851)
- Read more about DPARSFA 报错
- Log in or register to post comments
- 4457 reads
DynamicEC 报错
设置情况如下
win2008, Matlab 2011b
出错文件如下
Error using DynamicBC_sliding_window_FC>wgr_correl_FCD (line 220)
Error: The expression to the left of the equals sign is not a valid target for an assignment.
Error in DynamicBC_sliding_window_FC (line 41)
- Read more about DynamicEC 报错
- 6 comments
- Log in or register to post comments
- 11004 reads