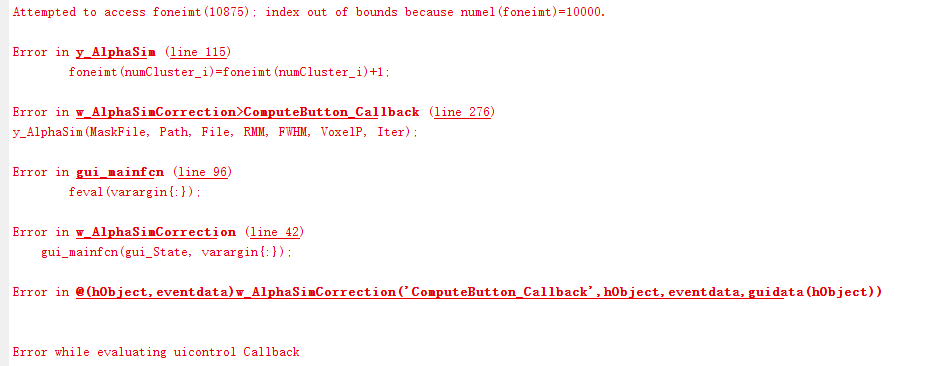
使用dparsf for monkey 版本处理猴子的脑数据,如何进行reorient
老师,您好!
我目前正在使用dparsf 4.0 for monkey data 版本处理静息态猴子脑数据,采用的参数设置如下(事先已经去除了前6个时间点):
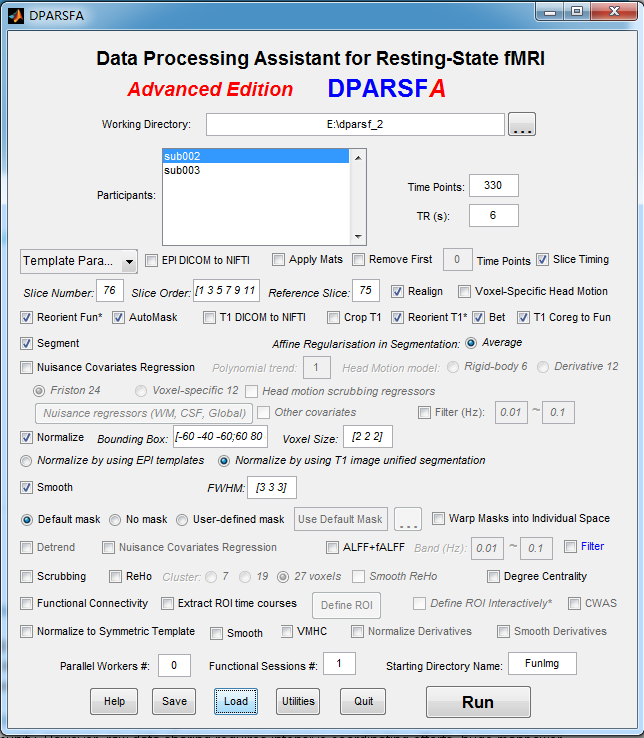
图1 参数设置
- Read more about 使用dparsf for monkey 版本处理猴子的脑数据,如何进行reorient
- 5 comments
- Log in or register to post comments
- 11457 reads