Submitted by seuzjj on Tue, 07/09/2013 - 10:38
各位老师,同学好,
请教一下,用DPARSFA处理fMRI时我使用了T1项做coregistration,然后做normalization,但我没找到normalize 到 MNI space之后的T1 MRI.
是不是说,用做完coregistration 之后的 T1 计算如何normalize fmri 到 MNI space后仅仅把参数用在fMRI上,并没有保存 MNI space中的的T1 ? 有没什么方法可以得到它呢?
另外在PicturesForChkNormalization 中有些subject 有些灰质和颅骨都重合了,如下图横断面,这样的subject是不是就不可以用了?或者是不是哪里的参数设置不合适,如何改进它呢?
非常感谢!
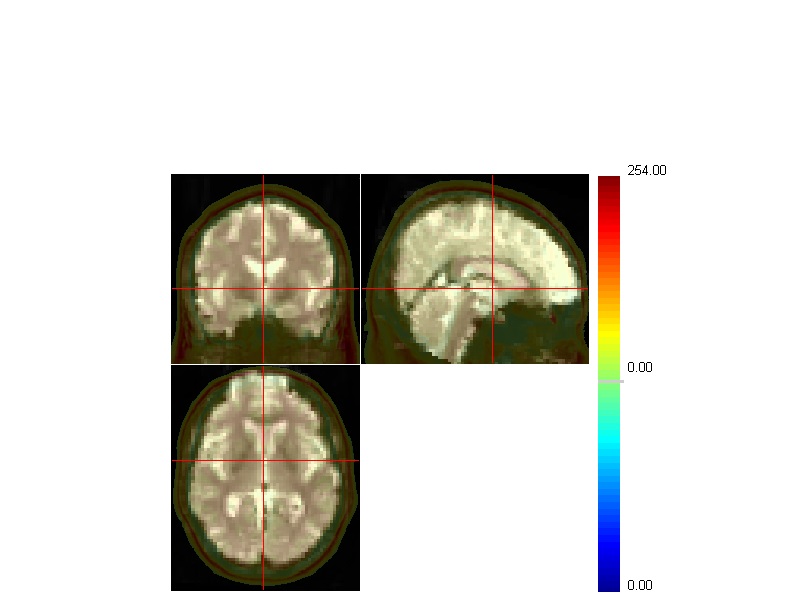

Submitted by ZangYF on Wed, 07/10/2013 - 05:57 Permalink
Re: 请教:如何得到MNI space 中的T1 MRI
我对具体操作不是特别精通。(1)coregister之后的3D-T1,好像只是头信息修改了。(2)这个图的配准效果的确有问题。我建议你略调整一下3D-T1的角度,然后再跑一遍,甚至不用调整,再跑一遍试试。据我所知,配准过程有一定的不确定性,跑两遍的结果并不完全相同。
Submitted by YAN Chao-Gan on Sat, 07/13/2013 - 05:25 Permalink
Re: 请教:如何得到MNI space 中的T1 MRI
在T1ImgNewSegment下面找到对应的结构像,但是加了一个前缀w,即为在MNI空间的结构像。
同意臧老师的看法,这个被试最后再做一些处理。有没reorient Fun* 和 reorient T1*?
Submitted by seuzjj on Mon, 07/15/2013 - 11:45 Permalink
Re: 请教:如何得到MNI space 中的T1 MRI
非常感谢两位老师的解答, 我在T1ImgNewSegment下找到了MNI空间的结构项。

我用了reorient Fun* 和 reorient T1*,重复了几次,配准效果一直不好,和上面贴图类似,是不是这样的就要排除了呢?
另外老师可否帮我确定下,先后做了slice timing, realign 和covariate regression的功能项是类似于下图的结果,这样是正常的结果么?
谢谢^_^
Submitted by ZangYF on Mon, 07/15/2013 - 14:02 Permalink
Re: 请教:如何得到MNI space 中的T1 MRI
我的回答只针对这个问题“我用了reorient Fun* 和 reorient T1*,重复了几次,配准效果一直不好,和上面贴图类似,是不是这样的就要排除了呢?”,其它问题我不太懂。
我觉得如果是病人数据,仍掉很可惜,可以考虑只用EPI配准,感觉比通过高清结构像成功机会大得多。当然,如果其它数据采用高清结构像,只有这一个被试采用EPI,可能会被质疑。
Submitted by seuzjj on Mon, 07/15/2013 - 18:48 Permalink
Re: 请教:如何得到MNI space 中的T1 MRI
谢谢臧老师的建议, 我觉得您说的是个很好的折中, 我会尝试的.
Submitted by YAN Chao-Gan on Wed, 07/17/2013 - 00:32 Permalink
Re: 请教:如何得到MNI space 中的T1 MRI
1. 核查哪一步出的问题。
1.1. 配准好的结构像效果如何?
1.2. 用两个MRIcroN分别打开T1ImgCoreg下的结构像,与Realigh parameters下的平均功能像,看coregistration的效果如何?我猜测是这一步的效果不好。我在想办法更好地解决一下这个问题,也许能在12月份发布一个新版本。
2. 正常。