Submitted by xyuan on Sun, 09/30/2012 - 22:19
学习vbm分析,使用了dparsfa的新分割与dartel的模板处理,非常方便,但是我想知道软件内部具体的操作流程与参数如何设置的?最后出来的结果应该是平滑过了,平滑核是多少?直接统计好像都是散点,我自己又平滑(8)一次;我自己又按vbm tutorial(john ashburner写的)的流程与参数跑了一遍,感觉出来结果不同,所以想知道贵软件vbm的流程参数设置如何,与vbm tutorial文中区别在哪里?还有贵软件做vbm过程中,dicom格式转换有问题,co前缀有时候缺失,软件就卡住了;我自己mricron转好,放入T1img,勾掉dicom转换,发现软件又不能跑下去;后来发现从NII文件开始,也要建立T1raw的文件夹,里面还得有相关文件夹(空的也行);是否也可按功能数据一样,选从哪步开始。
跑完数据后,smwc1前缀的文件是121*145*121,1.5*1.5*1.5,接下去是我最困惑的地方,我一直是用rest的,我是应该讲这批数据重采样成restBrainMask_05_61x73x61的3*3*3做下去?但是用rest工具里reslice image发现无法重采样一组数据,bug?于是我只好将mask重采样成1.5*1.5*1.5了,spm双样本分析好,想用sliceviewer看,提示this NifTi image includes an rotation transformation.You should reslice this data before attempting to overlay the image,由于自身对vbm一知半解,卡在这里,请老师指教,万分感谢!

Submitted by YAN Chao-Gan on Mon, 10/08/2012 - 23:44 Permalink
Re: vbm分析
不好意思,这个问题回复要比较长,我20号再回答。:)
如果到时候忘记了,请记得提醒我。
Submitted by xyuan on Sun, 10/21/2012 - 23:36 Permalink
Re: vbm分析
严老师,21号了,记得回复哦,谢谢!
Submitted by Fan Cx on Mon, 12/01/2014 - 17:18 Permalink
Re: vbm分析
严老师,我也遇到了上述问题,请问怎样解决?
Submitted by YAN Chao-Gan on Mon, 10/22/2012 - 10:50 Permalink
Re: vbm分析
1. 具体步骤:
(1) DICOM to NIFTI. (2) "New Segment + DARTEL": New Segment -- The structural image is then segmented into gray matter, white matter and cerebrospinal fluid by using "New Segment" in SPM8. (3) "New Segment + DARTEL": DARTEL -- Create Template, and DARTEL -- Normalize to MNI space (Many Subjects) for GM, WM, CSF and T1 Images (unmodulated, modulated and smoothed [8 8 8] kernel versions).
注意,此处三种版本(unmodulated, modulated and smoothed)都是由DARTEL (Normalize to MNI space)得到的。
2. 确保你所有的被试都同质,至少转出应该有以c开头的文件,这样是能够继续的。
3. 你可以转成NIFTI格式(注意,每个被试下面有且只有一个NIFTI结构像文件),放在T1Img下。
4. T1像不能选择从那一步开始。如果选了T1 DICOM to NIFTI,是从T1Raw开始,如果没选,是从T1Img开始。
5. 如果做VBM分析,为什么要把高精度的结构像重采样成低精度的3*3*3?
6. 双样本分析好 - 你是基于MNI空间的图像进行的吗?(如smw之类的)。如果是,应该不会有这样的提示。
Submitted by xyuan on Mon, 10/22/2012 - 22:59 Permalink
Re: vbm分析
谢谢严老师耐心回答,这些天自己也看了vbm tutorial,感觉应该是一样的,于是今天又重新跑了一遍,发现smwc1前缀文件(我理解是平滑过的灰质体积),打开看了下发现灰质是黑的,打开 swc1文件,灰质倒是正常的,亮的。太奇怪了!
Submitted by YAN Chao-Gan on Tue, 10/23/2012 - 01:44 Permalink
Re: vbm分析
黑的?是指什么?MRIcroN打开?
另外,不会自动生成swc1文件的。
Submitted by xyuan on Tue, 10/23/2012 - 21:11 Permalink
Re: vbm分析
跑完T1imgnewsegment文件夹里随便选个sub,里面有BiasField_co*.nii,C1-6co*.nii,co*_seg8,iy_co*.nii,mco*.nii,rc1-6co.nii,smwc1-3co*.nii,swc1-3co*.nii,u_rc1co*_Template,y_co*.nii这些文件,然后我用mricro打开,我理解smwc1是平滑后的灰质体积,灰质位置应该是白色的,而这里打开是黑色的,跟这个文件夹里其他c1相关的文件打开是相反的。第1副图白底黑边的是smwc1,其他c1图像都是类似第2副黑底白边(图2是swc1),这跟我理解的相反啊,跟vbm8跑出来的也是颜色相反的……。谢谢!
Submitted by YAN Chao-Gan on Wed, 10/24/2012 - 05:12 Permalink
Re: vbm分析
你左图是用什么软件打开的?MRIcroN打开背景不会是白色呀。把图片贴全一点,附上colorbar。
Submitted by xyuan on Wed, 10/24/2012 - 20:26 Permalink
Re: vbm分析
mricro,换mricron也一样;平时都是用mricro打开的,我也是第一次看到这样的;打开时候会跳图1的对话框,确定后如图2,;但是其他灰质图像都会跳图1的对话框。.jpg)
Submitted by xyuan on Wed, 10/24/2012 - 20:28 Permalink
Re: vbm分析
图1没有了,图1的对话框就是 this NifTi image includes an rotation transformation.You should reslice this data before attempting to overlay the image
Submitted by YAN Chao-Gan on Fri, 10/26/2012 - 01:54 Permalink
Re: vbm分析
不要用MRIcro打开NIFTI格式的数据。你再用MRIcroN打开截下图。
Submitted by xyuan on Sat, 10/27/2012 - 13:32 Permalink
Re: vbm分析
mricron打开没有提示对话框出现,但是图像还是一样的。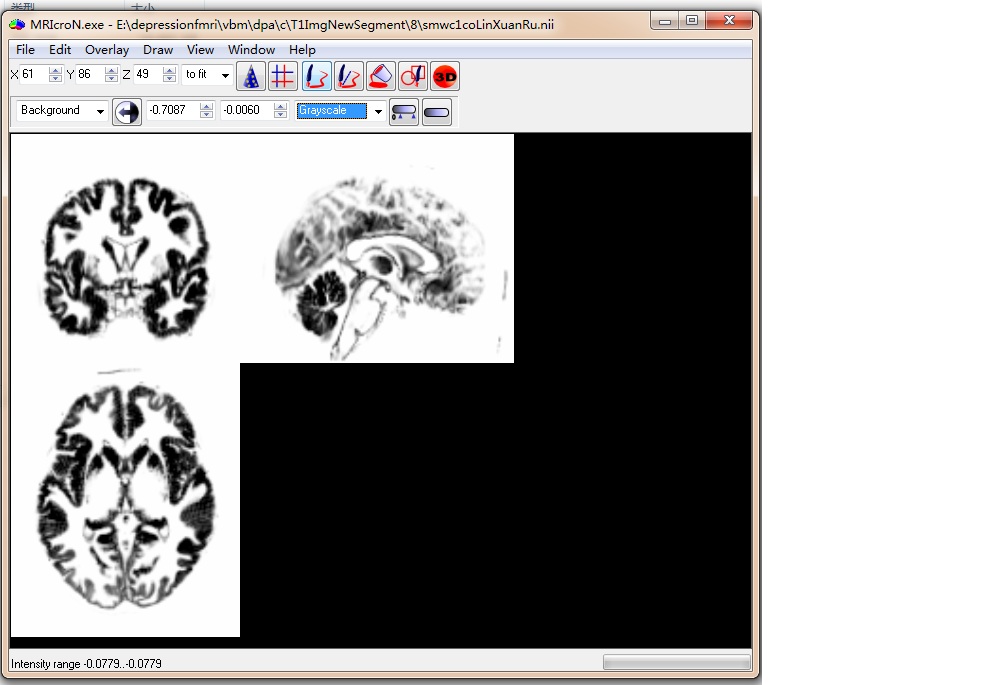
Submitted by YAN Chao-Gan on Mon, 10/29/2012 - 22:54 Permalink
Re: vbm分析
很奇怪,在灰质文件中不应该有负值。但你的这个图像里面都是负值。
你重做一次,或者看看别的被试?
Submitted by Fan Cx on Mon, 12/01/2014 - 17:16 Permalink
Re: vbm分析
我也遇到了同样的问题,请问您是怎么解决的?
Submitted by confirm on Mon, 03/17/2014 - 22:57 Permalink
Re: vbm分析
我的也提示没有CO*文件..
Submitted by ZHANG_RESTadmin on Wed, 03/26/2014 - 14:02 Permalink
Re: vbm分析
可以自己先用MRIcron的dcm2nii转成o和co前缀的数据。