Submitted by loveH on Thu, 12/18/2014 - 09:38
老师:
您好!
我用DPARSFA对静息态的数据进行预处理,顺序是:SliceTiming-Realign-T1Segment-Nuisance Covariates Regression-Mormalize.遇到了两个问题:
1.我发现Nuisance Covariates Regression的时候生成的某些被试的白质、灰质、脑脊液、全脑信号mask的坐标位置与realign得到的图像的坐标位置有差异。如图(左侧为realignParameter文件夹内的图像,右侧为Masks中BrainMask图像,用MRIcro显示的同一坐标位置的截图):
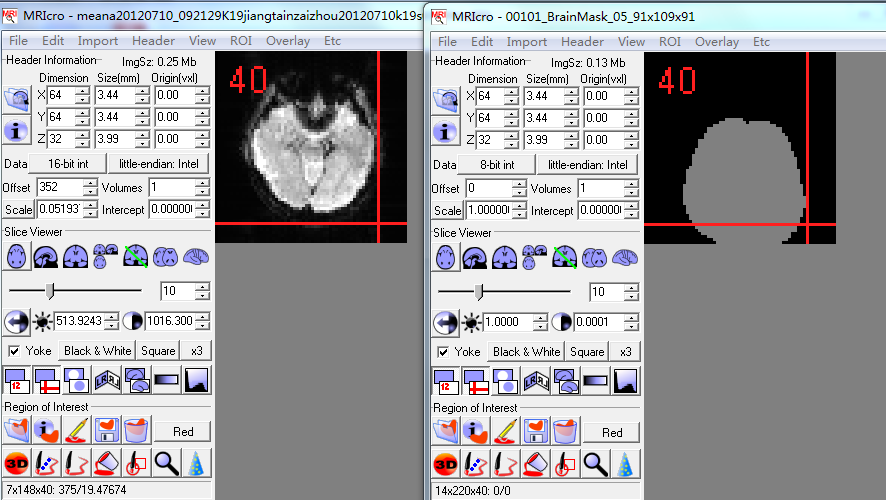
但是T1分割的结果看起来坐标位置是正常的。我在对同一个被试做一次相同步骤的预处理,得到的图像坐标位置就相同了,请问这是什么原因呢?
因为我的一批被试200人左右,其中有20个左右出现这个状况。对于这些坐标偏差的被试我用什么方式可以调整一下mask的位置呢?
2.有一些BrainMask在小脑位置最低层看起来会缺少一半,如图:
.png)
这样子的mask怎么处理呢?
谢谢老师~

Submitted by ZHANG_RESTadmin on Sat, 12/20/2014 - 17:02 Permalink
Re: DPARSFA回归WM,GM,CSF时生成的mask与Realign得到的图像的坐标不一致
这些mask文件都是标准空间的,而你的EPI图像却是个体空间(或者原始空间)的。这两个图像本来就不会匹配。
你要弄清楚你用mask提取信号的时候,是对什么图像做的提取,勾选normalize之后的nuisance covariate regression,就是对标准空间的EPI图像进行提取的,你应该先确保你勾对了位置,再比较模板和已经配准的EPI图像,不能用一个原始的EPI图像去和模板比较。
Submitted by loveH on Mon, 12/29/2014 - 16:50 Permalink
Re: DPARSFA回归WM,GM,CSF时生成的mask与Realign得到的图像的坐标不一致
好的 我明白您的意思 但是我的处理是基于个体空间的
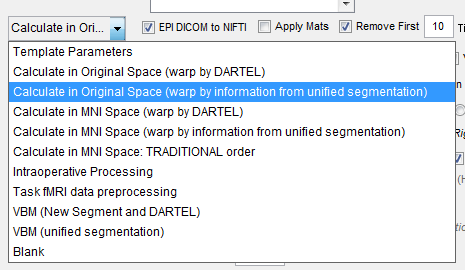
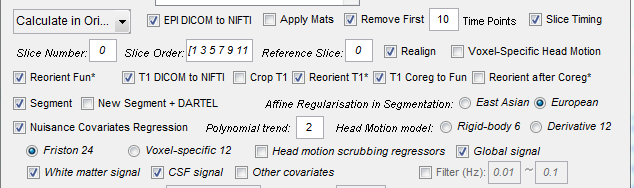
步骤的截图:
然后就会出现上面说的问题了……
那再请问老师一个问题:那在生成mask文件夹中的每个个体的WM,GM,CSF和全脑的mask的时候,是怎么计算的呢?使用spm的defprmations功能进行反配准得到的嘛?那请问使用的插值方式是trilinear还是其他呢?