Submitted by ZHANG_RESTadmin on Tue, 03/27/2012 - 11:04
My major research interest is Independent Component Analysis (ICA): methodology and applications.
(我主要的研究兴趣为ICA算法和应用)
If any questions regarding with ICA, please feel free to post here and I will try my best to solve them.
(因此,如果各位有任何独立成分分析ICA相关问题,请在这里提出,我每天会尽量回答并解决)
And I will post some helpful information about ICA each day if I have time.
(我也会贴出一些ICA相关知识在这里,尽量做到每天)
- ZHANG_RESTadmin's blog
- Log in or register to post comments
- 165709 reads

Comments
Submitted by liufeng on Tue, 04/17/2012 - 10:57 Permalink
期待张师兄贴出一些关于ica的知识
期待张师兄贴出一些关于ica的知识
Submitted by linxiao on Sat, 09/21/2013 - 16:07 Permalink
Re: 期待张师兄贴出一些关于ica的知识
老师好,请问一下您有没有默认网络的模板啊,能不能发我QQ一下啊,1039876318万分感谢!!!
Submitted by ZHANG_RESTadmin on Mon, 09/23/2013 - 12:31 Permalink
Re: 期待张师兄贴出一些关于ica的知识
已发qq邮箱
Submitted by lipingchxyy on Tue, 07/01/2014 - 10:58 Permalink
Re: 期待张师兄贴出一些关于ica的知识
张老师您好:
用什么软件进行 ICA 分析,我看有的研究者用GIFT软件。您那里用什么软件进行ICA分析?
另外,你能发一些关于ICA的知识吗?我的邮箱是:lipingchxyy@163.com.
期待您的回复!
Submitted by ZHANG_RESTadmin on Tue, 07/01/2014 - 14:02 Permalink
Re: 期待张师兄贴出一些关于ica的知识
GIFT, FSL MELODIC, MICA 都可以的!
Submitted by yeshion on Sun, 07/13/2014 - 22:29 Permalink
Re: 期待张师兄贴出一些关于ica的知识
张老师好:
我利用MICA软件提取了DMN网络。现在观察患者每个DMN 的功能连接异常区域是否和患者预后评分有关系。对于每个DMN ,先在双样本t- 检验的结果中提取出患者相比正常对照降低的脑区(P<0.05,AlphaSim校正),作为ROIs。然后应用REST里的Extract ROI Signals工具提取ROI Singals,但是运行时出现以下错误提示,小弟非工科,看不懂代码,恳请老师指点,不胜感激!
forum/sites/default/files/users/yeshion/image/QQ%E5%9B%BE%E7%89%8720140710192036.jpg
Submitted by ZHANG_RESTadmin on Mon, 07/14/2014 - 15:32 Permalink
Re: 期待张师兄贴出一些关于ica的知识
你仔细看看你生成的ROI文件,是不是用MRIcron看看他们voxel上的值会出现0和1以外的数字,
另外,你的roi文件为何是.nii.img后缀?请check一下到底是什么?如果是img,看看与之相对应的hdr文件有没有。
另外,你应该说下你的ROI怎么提出来的。具体步骤,我好帮你分析错误。
Submitted by yeshion on Tue, 07/15/2014 - 21:56 Permalink
Re: 期待张师兄贴出一些关于ica的知识
非常感谢张老师的回复。
1.用REST软件统计分析(双样本t检验)患病组和正常对照组具有显著差异性的脑区,把这些差异性脑区(存活的clusters)在REST Slice viewer里保存下来(save cluster),这就是我的ROI,
2.用REST Slice viewer保存cluster生成的ROI文件是NII文件,没有对应的hdr文件
3.用MRIcron打开这些ROI,可以看到这些clusters,但不知道怎么看voxel的值,我把ROI文件发到您的邮箱了(上传不了这个论坛),望查收!
期待您的回复!
Submitted by ZHANG_RESTadmin on Wed, 07/16/2014 - 11:54 Permalink
Re: 期待张师兄贴出一些关于ica的知识
见我给你的回复。
首先,你应该用REST的one sample t test分析患者组的DMN,得到一个单样本显著的DMN,
Submitted by yeshion on Wed, 07/16/2014 - 17:52 Permalink
Re: 期待张师兄贴出一些关于ica的知识
Submitted by ZHANG_RESTadmin on Thu, 07/17/2014 - 23:30 Permalink
Re: 期待张师兄贴出一些关于ica的知识
一般单样本t检验结果经得起校正。如果经不起,可能你样本量小,或者得到的成分不好。
是的,具体表示这个网络在这个ROI上的功能连接值。可以做相关。
Submitted by yeshion on Fri, 07/18/2014 - 00:22 Permalink
Re: 期待张师兄贴出一些关于ica的知识
谢谢张老师的回复!
我再请教您一个问题
用REST roi signal extraction提取后的txt文件内容代表这个网络在这个ROI上的功能连接值。按照前人的理论,连接值越低,患者的预后评分肯定不好的。可是我发现连接值低的却 预后好呢? 而且发现正常人的功能连接值都不是很高。难道这些数值还需要转换吗? 还是有可能是其他问题?
Submitted by ZHANG_RESTadmin on Sat, 07/19/2014 - 17:38 Permalink
Re: 期待张师兄贴出一些关于ica的知识
连接值在前人研究里面,也是ICA得到的值吗?还是种子点FC得到的?两个略有不一样,不能平推过来。
至于相关方向错了 我觉得也不一定是错,关键你怎么解释。
Submitted by lipingchxyy on Sat, 08/30/2014 - 00:40 Permalink
ICA相关知识
张老师您好:
有几个问题想请教您:
1.在对静息态的FMRI数据进行ICA分析时,FSL MELODIC 软件和MICA软件,其分析的结果有什么差异吗?
2. 我仔细阅读了您发给我的课件,以及MICA的操作流程。每个被试在进行ICA分析后,有很多的成分,我们怎么能确定这些成分的性质呢?(如这个是ACC的成分,那个是OFC的成分?)
3.如果我想在独立成分分析的基础上,进行功能连接的分析(每个独立成分挑选出来,在这些成分之间做功能连接)。我该怎么做呢?
非常感谢张老师的帮助!期待您的回复!
Ping
Submitted by Duoning on Fri, 05/08/2015 - 10:47 Permalink
Re: ICA相关知识
请问这个问题你解决了吗?
Submitted by 杜肖 on Fri, 09/25/2020 - 18:32 Permalink
Re: 期待张师兄贴出一些关于ica的知识
请问一下,提取ROI时间信号,输入的文件夹是什么数据啊,是经过smooth之后4DfMRI数据还是ICA之后我挑出来的(如默认网络)各个被试的这个成分
Submitted by Duoning on Tue, 04/07/2015 - 19:56 Permalink
Re: 期待张师兄贴出一些关于ica的知识
请问MICA中是如何提取出DMN的?
Submitted by ZHANG_RESTadmin on Tue, 04/07/2015 - 20:47 Permalink
Re: 期待张师兄贴出一些关于ica的知识
DMN要通过肉眼挑出来。确定要成分的序号以后,再通过extract功能把所有人的该成分提取出来用于统计分析。
Submitted by yeshion on Sat, 06/28/2014 - 12:42 Permalink
Re: 期待张师兄贴出一些关于ica的知识
这位兄弟,张老师发给你的默认网络的模板,能给我发一份吗?谢谢啦!
yeshion2012@126.com
Submitted by Duoning on Tue, 01/13/2015 - 11:30 Permalink
Re: 期待张师兄贴出一些关于ica的知识
同求默认网络的模板,谢谢。邮箱:csu_duoning@163.com
Submitted by Harry on Wed, 08/05/2015 - 09:18 Permalink
Re: 期待张师兄贴出一些关于ica的知识
您好!求DMN的mask+1.。。不知道您拿到了没?可否发我一份?跪谢!thuwangkai12@sina.com
Submitted by Miki on Fri, 07/27/2012 - 23:50 Permalink
choice of preprocessed images for ICA
Hi, I learn that we can input FunImgARWS images in ICA analysis using MICA, but I also read that some studies applied detrend and filter to the FunImgARWS images before inputting the preprocessed images into ICA. Does it matter much between the choice of preprocessed images (FunImgARWS or FunImgARWSDF) for ICA? Thanks!
Submitted by ZHANG_RESTadmin on Wed, 08/01/2012 - 09:30 Permalink
Yes, some papers used
Yes, some papers used smoothed data for ICA, while some did use the detrend or filtered data for ICA.
No systematical comparision between the two preprocessing methods in ICA studies.
I recommand the first one.
Submitted by paolozanini on Tue, 04/16/2013 - 14:55 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
Hi!
I have a question regarding ICA.
I'm using the fastICA algorithm and I have this problem. My two dependent variables, say X1 and X2 are mixtures of one common non gaussian variable T and two gaussian error terms. My algorithm is able to recover the signal T (I did a lot of simulations), but I want to prove it theoretically. Do you have any suggestion?
My model can be written as:
X1=T+E1
X2=T+E2
where T is non gaussian, E1 and E2 are gaussian and the three variables (T, E1 and E2) are independent.
Thank you in advance for your help
Submitted by ZHANG_RESTadmin on Wed, 04/17/2013 - 12:47 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
Hi, can you post your question more clearly? So we can discuss more deeply.
As far as I know, fastICA model is not like X1 = T+E1. So I assumed that X1, X2 are the variables you got from fastICA, and that you intended to further decompose those variables into mixture of a nongaussian and a gaussian additive variable. Did I understand correctly?
The general rule is if E1 and E2 are two independent variables with Gaussian distribution, they can not be separated by using ICA.
Han
Submitted by Chen0075 on Wed, 05/08/2013 - 22:04 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
Hi, Dr. Zhang,
I have a question about ICA. Is it reasonable to perform ICA in follow-up study, since a subject's data at baseline and follow-up time are interelated each other。
Best Wish!
Submitted by liuchen on Sun, 05/26/2013 - 23:20 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
我在使用mica的时候,在成分挑选的时候,gui view界面中点击sort components,并且选择空间相关并加载了需要的DMN网络模板,点击ok之后,软件没有任何反应,
问题1:恳请张寒老师指导一下,mica中sort components的使用。
问题2:identify components在使用默认的设置,全部勾选,点击ok,matlaB就会报错,请问identify components的作用以及他的使用方法。
Idendentify components
??? Undefined function or variable 'dim'.
Error in ==> view_gui>ButtIdenComp_Callback at 898
[ICNOs]=nic_removeNoiseComponent(compNum,dim,outPrefix,threshold,zipFilePath,a);
Error in ==> gui_mainfcn at 96
feval(varargin{:});
Error in ==> view_gui at 30
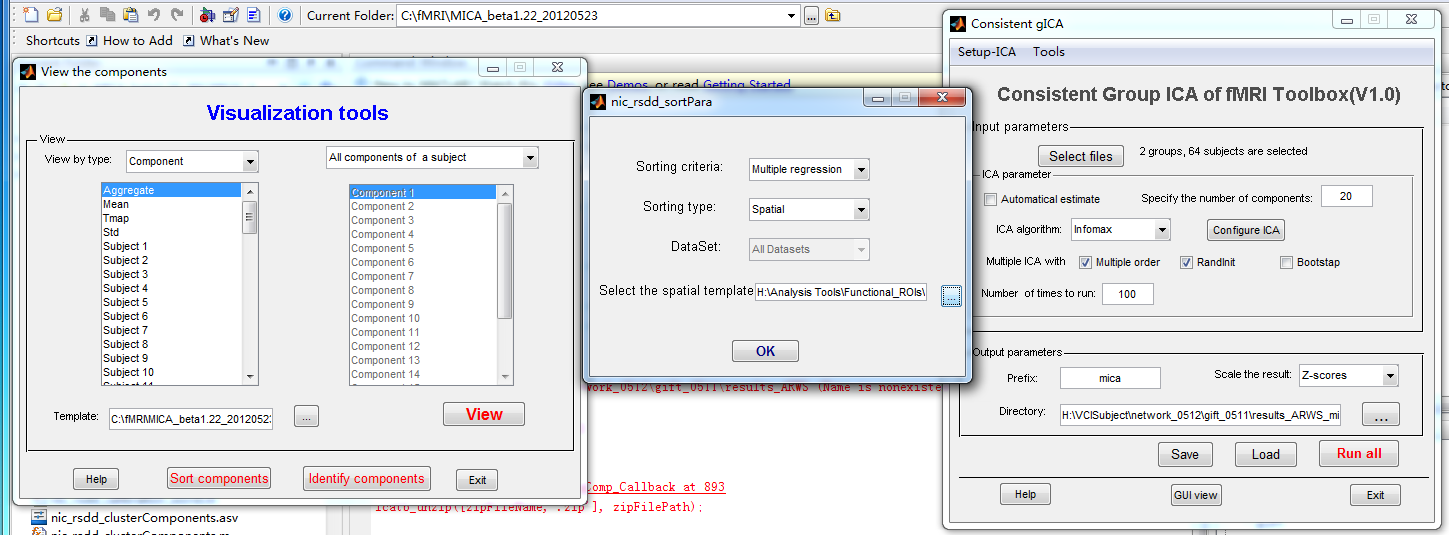
gui_mainfcn(gui_State, varargin{:});
??? Error while evaluating uicontrol Callback
Submitted by liuchen on Wed, 07/03/2013 - 16:21 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
望大家帮忙回复一下
Submitted by Duoning on Tue, 01/13/2015 - 11:28 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
我现在遇到了同样的问题,请教你的问题是如何解决的?期待你的回复,QQ:1020325646
Submitted by linxiao on Mon, 09/16/2013 - 19:55 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
老师好:
关于独立成分分析还是有些地方不明白,在成分提取之后,如何对已提取的成分进行筛选,看文章里面说筛选步骤主要分两步,(1)首先由于背侧注意网络的频带属于低频段(0.01-0.1Hz),分析各个独立成分对应的时间序列的功率谱,如果高频信号(>0.1Hz)占该独立成分的功率谱 50%以上,则去除该成分.功率图谱是怎么看的呢???(2)然后在剩余的成分中用一个模板匹配程序选择与背侧注意网络模板最匹配的成分,该模板匹配程序主要是计算每个成分中落在模板内体素的 z 值的平均值与模板外体素的 z 值的均值之差,定义为拟合度值(goodness-of-fit scores),选择拟合度值最大的成分作为最匹配(best-fit)的成分,即可认为该成分是背侧注意网络。请问老师,这两步具体是怎么实现的呢??希望能得到老师指点…………
Submitted by Nicole on Wed, 04/22/2020 - 11:47 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
我也有同样问题
Submitted by 18483620297 on Sun, 06/14/2020 - 13:53 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
你好,我想问一下,我下载MICA toolbox 后,在matlab里面也设置了路径,但是在matlab 命令空间输入mica 的时候报错。(如下所示)是怎么回事。你可以把你mica toolbox 给我一份吗? 谢谢!
Error using fileparts
Too many output arguments.
Error in nic_rsdd_misc (line 15)
[pathstr, name, ext, versn] = fileparts(mfilename('fullpath'));
Error in mica (line 13)
pat=nic_rsdd_misc('whereis');
Submitted by 杜肖 on Fri, 09/25/2020 - 16:36 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
同学我知道在哪看功率谱,但是关于ICA后续的一些问题我一窍不通,这是我的QQ420074030,希望可以多多交流
Submitted by joaquincuomo on Wed, 03/19/2014 - 16:34 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
Hi,
I'm trying to use ICA to denoise an EEG recording, but first I checked how ICA works.
I tried decomposing a sin + sawtooth + white noise, which works wonderful. But when I added the same signals with delays it stopped working (according with my readings that's Time Embedded Data, which should improve the performance). The same result when I repeated the inputs varying only amplitudes (without any delay). Am I doing something wrong?
I attached a piece of code down here to illustrate my problem.
N = 5000;
Fs = 500;
t = (0:(N-1))/Fs;
% 3 independent signals
a = sin(t*2*pi);
b = sawtooth(t*2*pi*5);
c = rand(1,N);
% 3 signal built mixing linearly the independent signals
x1 = zeros(N,3);
x1(:,1) = rand*a+rand*b+rand*c;
x1(:,2) = rand*a+rand*b+rand*c;
x1(:,3) = rand*a+rand*b+rand*c;
% a bigger set of signal usign x1 with delays
delay = 5;
x2 = x1(delay*1:end,:);
x3 = x1(delay*2:end,:);
x4 = x1(delay*3:end,:);
x5 = x1(delay*4:end,:);
Lm = length(x5);
x = [x1(1:Lm,:) x2(1:Lm,:) x3(1:Lm,:) x4(1:Lm,:) x5];
x = x';
[Y,W,P] = ica(x);
Thanks!
Submitted by ZHANG_RESTadmin on Wed, 03/26/2014 - 13:45 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
i hav no idea which ica algorithm you used in the script, but in my opinion you failed because the signals look all the same (some signals can not be decomposed). the time delay is too small to be differentiated from others.
Submitted by yeshion on Thu, 09/04/2014 - 15:37 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
张教授:
您好!
我再应用MICA时,数据跑到最后出现以下问题(我的参数:病人15例正常人15例,经过slice timing、realign、normalization,smooth,一共30个被试放在one group在MICA里跑,成分数选择20个,Z-score转换,其他都是默认状态)
forum/sites/default/files/users/yeshion/image/IMG_20140903_225351.jpg
Submitted by wujiexiaoxiao on Wed, 05/25/2016 - 17:40 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
张教授:
您好!在用ICA跑正常人和病人的数据的时候,成分的双样本差异结果随着成分数的不同发生变化,请问您是如何处理这类问题的?我该怎么样选择成分数?以及这样选择的依据是什么?
期待您的回复,祝好!
Submitted by Duoning on Wed, 12/31/2014 - 15:55 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
MATLAB运行mica第一次报错如下:
退出,load参数设置的mat文件,运行有终止了,报错如下:
Exception occured. (MATLAB:innerdim)
矩阵维度不同,请问是什么原因?。是两组数据,第一组和第二组被试人数不同
如果MICA终止过一次,下次再运行,是不是所有步骤都要重新来过?
Submitted by niuwen on Sun, 05/10/2015 - 22:16 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
STARTING DATA REDUCTION (PRINCIPAL COMPONENTS ANALYSIS)
Submitted by wujiexiaoxiao on Sat, 10/10/2015 - 17:08 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
关于ICA算法的问题
老师您好,看了calhoun的2001年(A Method for Making Group Inferences from
Component Analysis)和2009年的文章(A review of group ICA for fMRI data and ICA for joint inference of imaging, genetic,
and ERP data),有如下几个问题:
1.我的理解是对于每个个体group ICA GIFT软件back reconstruct 出来的同一个成分的map是不一样的,但是同一个成分的time course 是一样的并且和整个group的是一样的。我的理解对吗?老师
2. 2009年的文章中说time concatenation时是这样说的:The temporal concatenation approach allows for
里面说的unique time courses指的是什么,common group maps 指的又是什么,希望老师解答,谢谢老师!
Submitted by yubing on Fri, 05/27/2016 - 11:23 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
请教张老师,ICA分离出来的不同成分(默认网络、中央执行网络、额顶网络等等),有比较一致的模板吗?多谢!
Submitted by 18483620297 on Sat, 06/13/2020 - 19:53 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
大家好,
我是一名MICA的初级使用者,通过论坛我学会了一部分的操作。但是在run后出现了这样的错误。这是什么导致的呢?
有人能发我一份默认网络、运动网络、执行控制网络、记忆网络这四个网络的模板吗?(我想通过MICA提取出这四个网络的体空间和时间序列)
谢谢!
......................................
Group ICA Error Information:
Error using nic_spm_write_vol (line 20)
Inic_ompatible dimensions.
Error in ==> nic_spm_write_vol at 20
Error in ==> nic_write_vol at 37
Error in ==> nic_icatb_saveICAData at 117
Error in ==> nic_rsdd_groupStats at 178
Error in ==> nic_rsdd_runAnalysis at 201
Error in ==> RulAll_Callback at 473
Error in ==> gui_mainfcn at 96
Error in ==> moica_gui at 57
......................................
Exception occured. ()
Error using icatb_displayErrorMsg (line 23)
23#line, icatb_displayErrorMsg, in "E:\MICA\MICA_beta1.22_20120523\nic_functions\nic_icatb_helper_functions\icatb_displayErrorMsg.m"
44#line, nic_write_vol, in "E:\MICA\MICA_beta1.22_20120523\nic_functions\nic_spm5_files\nic_write_vol.m"
117#line, nic_icatb_saveICAData, in "E:\MICA\MICA_beta1.22_20120523\nic_functions\nic_icatb_saveICAData.m"
178#line, nic_rsdd_groupStats, in "E:\MICA\MICA_beta1.22_20120523\nic_rsdd_groupStats.m"
201#line, nic_rsdd_runAnalysis, in "E:\MICA\MICA_beta1.22_20120523\nic_rsdd_runAnalysis.m"
473#line, RulAll_Callback, in "E:\MICA\MICA_beta1.22_20120523\moica_gui.m"
96#line, gui_mainfcn, in "D:\matlab2012a_install\toolbox\matlab\guide\gui_mainfcn.m"
57#line, moica_gui, in "E:\MICA\MICA_beta1.22_20120523\moica_gui.m"
Error using fileparts
Too many output arguments.
Error in nic_rsdd_misc (line 15)
[pathstr, name, ext, versn] = fileparts(mfilename('fullpath'));
Error in view_gui>view_gui_OpeningFcn (line 76)
set(handles.templateFile,'String',fullfile(nic_rsdd_misc('WhereIs'),'template','nsingle_subj_T1_2_2_5.nii'));
Error in gui_mainfcn (line 221)
feval(gui_State.gui_OpeningFcn, gui_hFigure, [], guidata(gui_hFigure), varargin{:});
Error in view_gui (line 30)
gui_mainfcn(gui_State, varargin{:});
Error in moica_gui>guiView_Callback (line 656)
view_gui(handles.moicaCfg);
Error in gui_mainfcn (line 96)
feval(varargin{:});
Error in moica_gui (line 57)
gui_mainfcn(gui_State, varargin{:});
Error while evaluating uicontrol Callback
Submitted by 18483620297 on Sun, 06/14/2020 - 15:25 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
hello,我下载的MICA似乎有什么问题,大家能够给我一份可以完整运行的MICA toolbox 吗? wyang02960297@163.com
谢谢!
Submitted by 杜肖 on Fri, 09/18/2020 - 21:59 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
张老师你好,我最近想做ADHD_index评分跟ICA之后两组样本差异脑区的相关分析,我提取了八个静息态网络,然后做了双样本t检验(多重比较校正),我用这个作为提取roi的mask,然后把两组人放在同一个文件夹下提取roi时间信号,这样做对吗?非常希望您能帮我解答这个问题。
Submitted by 杜肖 on Fri, 09/18/2020 - 22:02 Permalink
Re: Questions about ICA plz posting here (独立成分分析相关问题我在这里做解答)
还有老师,如何提取这些roi内平均的体素z值呢