我做双样本T检验的mask做法对吗,谢谢!!!
各位老师:
我想咨询一下双样本T检验的mask问题, 我是MRI新手,我的感受是Rest软件最大的困难对我来说是每一个统计都让你选mask,而我不会制作mask,不点mask总觉得分析不规范,为此我为了mask摸索了一个月, 才敢用此软件。而SPM的分析只是点击,选默认mask。我根据严老师讲课视频和论坛中有老师写的"先用两组单样本T检验显著的脑区,在RESTimage Calculator:中根据公式(abs(i1)> threshold)+(abs(i2)> threshold)>0取他们的并集做一个mask供双样本检验用,这样小一点的mask才有可能使有些信息活下来的"这个原则,我进行了mask制作.但我不知道我做得对不对:
1. 我用DPARS算出两组各个人的smRehoMap等, 先对两组分别进行smReho-1 MAP单样本的T检验,因我没有假设,所以这步mask 我选的Rest软件自带的brain61X72X61模板作为mask(因我看文章中阳性区域报的结果太多了,尽管基底节更多一些), 然后, 单样本T检验中得到病人和对照组各自的T图,下一步,取两组T检验显著的脑区,先去看threshold value, 用RestSlice Viewer中将病人T图呈现出来, 我选择P=0.05, 得出Threshold=2.1448,又将对照组T图呈现出来, 选择P=0.05,得出Thresthold=2.093.有了threshold, 可以在Rest Image Calculator中根据公式: (abs(i1)> threshold)+(abs(i2)> threshold)>0, 输入(abs(i1)> 2.1448)+(abs(i2)> 2.093)>0, 出来的mask是个红色图(见图1,光盘映像文件,需用Rest Sliceview打开),我不知道这个过程或公式对否, abs是什么意思,是否不要加?
2. 于是, 我去掉abs, 又用(i1> 2.1448)+(i2> 2.093)>0(我看视频上为(i1>1.96+i2>1.96)>0)公式,出来的图比刚才那个图少些脑组织(见图2),不知道哪个对?
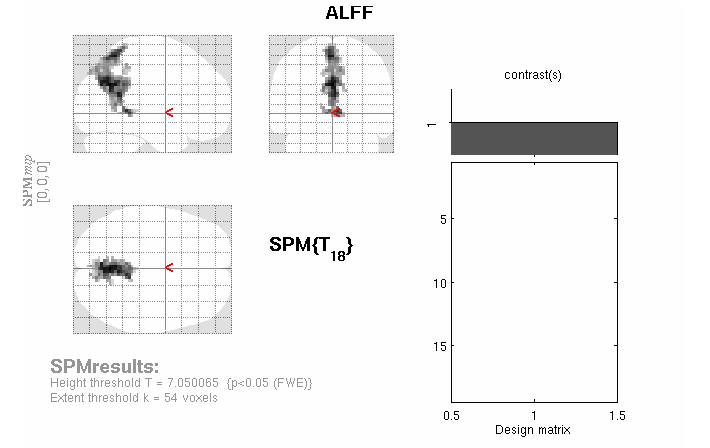
3. 最后我用第一个公式做出来的mask,作双样本T检验, 结果统计出来的下面的T图(见图3),但不知道是什么意思,这是 smReho 最后分析出来的是结果吗,我选P=0.05,cluster size=85, rmm=4,选了个图像裁了下来(见图4)?
4. 另外一个问题是如果不用大脑或灰质模板做mask的话,是否每做一个变量都要做mask,如分析mReho,ALFF, mALFF,fALFF等要按上述方法制作阳性结果并集的mask吗?
请各位能给予回复和指导,谢谢~~~~
- Read more about 我做双样本T检验的mask做法对吗,谢谢!!!
- 1 comment
- Log in or register to post comments
- 9366 reads