请教严老师及大家,如何提取杏仁核作ROI?谢谢
大家好,严老师好,我想比较两组被试的杏仁核体积、活动强度及功能连接强度的差异,除了手画、任务态激活区检测之外,现在比较常用的方法是怎样的,要用什么软件?期待回复
- Read more about 请教严老师及大家,如何提取杏仁核作ROI?谢谢
- 2 comments
- Log in or register to post comments
- 7659 reads
大家好,严老师好,我想比较两组被试的杏仁核体积、活动强度及功能连接强度的差异,除了手画、任务态激活区检测之外,现在比较常用的方法是怎样的,要用什么软件?期待回复
大家好:
我用REST-Utilities-XJVIEW-CI. Report计算cluster中VOXEL数目,发现Total的cluster数目是39,但是其中有WHITE MATTER 11个,这种情况下,在文章报道中,需要写39还是28?
谢谢
大家好:
在用dparsf软件得到ReHo,ALFF后,我用rest做了双样本t检验(对照组的smReHo,抑郁组的smReHo。对照组smALFF,抑郁组的smALFF)我有几个问题想问问大家。
1 这样做出来的结果具有统计学上的显著差异么? 如果没有请问为什么呢?
严老师您好:
想请教下Restv1.8中的Degree Centrality如何使用?Input data 取预处理后哪一步的数据,r 的threshold 该取多少合适?
Dear all,
我用zReHo,zfALFF做完单样本t检验,用Rest的Viewer看时,没办法调节p值,出现错误
??? Undefined function or method 'tcdf' for input arguments of
type 'double'.
大家好:
在rest做完双样本T检验后,我用MRIcro软件打开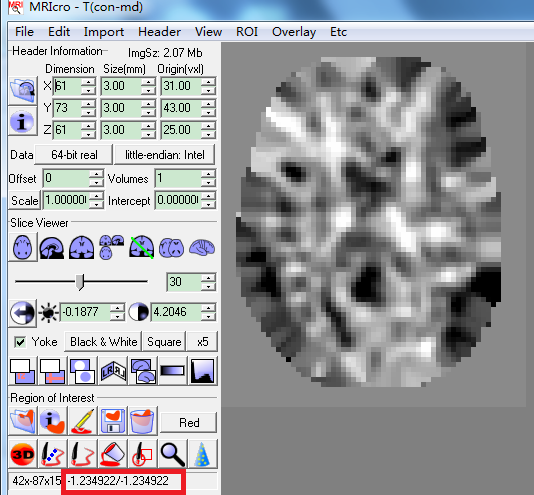 如图所示(红色标记) 我想知道这个比值代表这什么,有什么意义。
如图所示(红色标记) 我想知道这个比值代表这什么,有什么意义。
Dear all -
I extracted the connectivity matrix between all ROIs from the atlases provided with DPARSFA (AAL, Craddock, Dosenbach, ...). How do I find out the name of each ROI for each atlas? For the Harvard Oxford Atlas a file with anatomical labels is provided, but I'm not sure on how to use that.
各位好:
我用最近版的dparsf和rest跑数据,自动把我的数据存成了4D文件,这样导致我做回归全脑信号、wm csf信号时出错。而且貌似waitbar不出现了。
能不能自动的将4D convert成3D+time points呢?
谢谢