请问静息结构等数据的分析方法哪些是属于假设驱动哪些是数据驱动呢
我只知道ICA是数据驱动,那ALFF,Reho,FC还有VBM等等呢,需要先有假设吗?谢谢各位老师
- Read more about 请问静息结构等数据的分析方法哪些是属于假设驱动哪些是数据驱动呢
- 1 comment
- Log in or register to post comments
- 5352 reads
我只知道ICA是数据驱动,那ALFF,Reho,FC还有VBM等等呢,需要先有假设吗?谢谢各位老师
查了很多文献,一直搞不懂这两者什么关系,请各位老师指点
老师们好!
目前想利用ICA研究猴脑DMN,请问老师们知道猴脑DMN具体包括哪些脑区么?ICA得到的DMN成分,如何知道有哪些区域呢?
如想要比较两组猴子的DMN差异,是比较DMN网络分布图的差异,还是功能连接差异呢?采用什么工具包以及方法比较好呢? 求指教
承珍
如图,老的版本中FWHM只需填写自己用的平滑核是多大的就行了,今天用的发现旁边多出来个estimate,运行完后它好像可以自动把FWHM后面的空给填了,但完全不是自己用的平滑核的大小,想请问一下这个estimate的作用是什么? PS:用estimate填的FWHM后计算出的表,跟自己填的实际的FWHM结果不一样?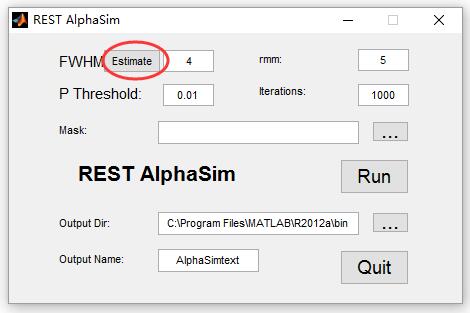
老師好,
目前的研究想要比較fALFF和ReHo兩種分析方法出來的結果相同與相異處,
想將兩個.nii的activation結果檔案呈現在同一張圖上(並標註為不同顏色),
但市面上的visualization software一次都只能放入一個.nii檔作檢視(ex: BrainNet Viewer等)
想請問這樣的問題各位使用者及老師是否有些建議或經驗可以分享
感謝萬分!
目前,正在使用dparsf处理猴脑数据,但是,采集的数据中存在大量的非脑组织(脖子以及颅外组织),最后配准到标准空间非常不理想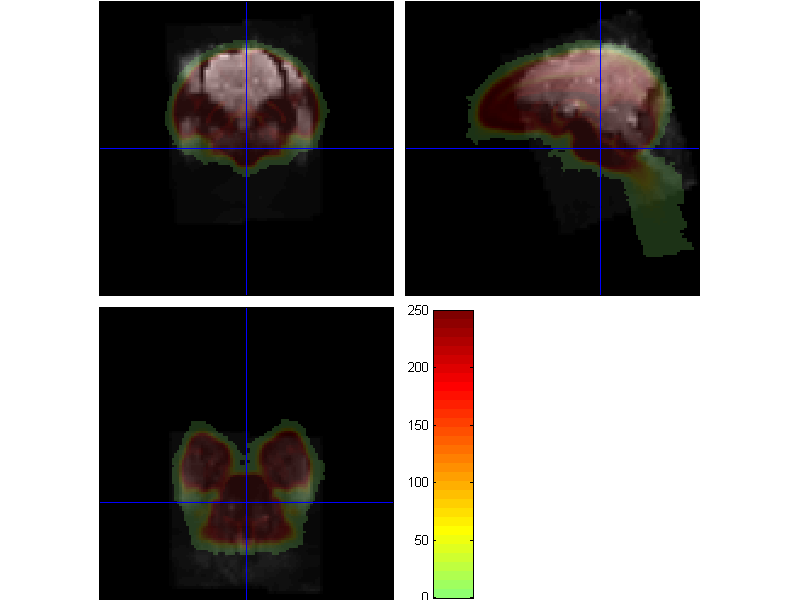 ,配准查看结果如下。如何提取脑组织啊,本人尝试用了fsl软件中的BET工具提取脑组织,但是结果不太理想,似乎去除了脑周围的部分,但是脖颈仍然未去除。请问,老师们怎么成功提取脑组织部分呢?
,配准查看结果如下。如何提取脑组织啊,本人尝试用了fsl软件中的BET工具提取脑组织,但是结果不太理想,似乎去除了脑周围的部分,但是脖颈仍然未去除。请问,老师们怎么成功提取脑组织部分呢?
你好,我目前正在研究两组数据间的差异。打算比较1.5T和3.0T不同机器扫描的数据间差异,新手一枚,望多指教!!!
存在几个问题,也顺便想问问楼主。
1、1.5T机器扫描的被试有10个,3.0T扫描的被试是另外的10subject。若想比较DMN的差异,是否有意义?
2、使用gift软件若要进行组独立成分分析group ICA,如何对这两组不同被试的数据进行group ICA呢? 目前,我只知道若是同样的10个subject的话,可以在数据输入的时候 选择2 session of 10subject。最后生成所有数据的mean component, 那要是1.5T和3.0T扫描的10个被试不同的要怎么进行group ICA呢?又如何进行DMN比较呢?一般是比较采用什么统计方法对何种差异进行比较?
第十四届多模态脑成像技术培训班
(基础和临床多模态脑成像技术专修班)
2016.12.2 – 2016.12.4
中国·北京
联合主办单位:
美德医疗
中国科学院生物物理研究所北京磁共振脑成像中心
支持协会:
中国认知科学学会
讲师单位:
北京大学磁共振成像研究中心
MD Anderson Cancer Center, Houston,Texas, US
一直用matlab2012b,用rest一直没问题,最近升级了matlab2015b,rest一直报错. 首先是matlabpool已经被移除了
Software Engineer
Job description
We seek a programmer who is an expert in C++ to join the Computational Functional Anatomy Laboratory at National University of Singapore. Our laboratory works on large neuroimage analysis. Your job responsibilities include but are not limited to, basic neuroimage processing, optimizing existing codes, writing Linux shell scripts to connect all image processes, and potential development of new medical image analysis tools. We offer high and comparable salary with industry.
Requirements: