北师大多模态神经影像与脑连接组学专题培训班开始报名了
大家好,
- Read more about 北师大多模态神经影像与脑连接组学专题培训班开始报名了
- Log in or register to post comments
- 4001 reads
大家好,
各位老师好!
我在用rest对一组mALFF做单样本t检验得到mALFF Tmap, 然后用slive viewer进行查看 得到的图像里面有负值,采用的FDR双尾矫正,Q值为0.001,单样本T检验出现负值
查看显著脑区不是应该只有正值吗 怎么会出现负值呢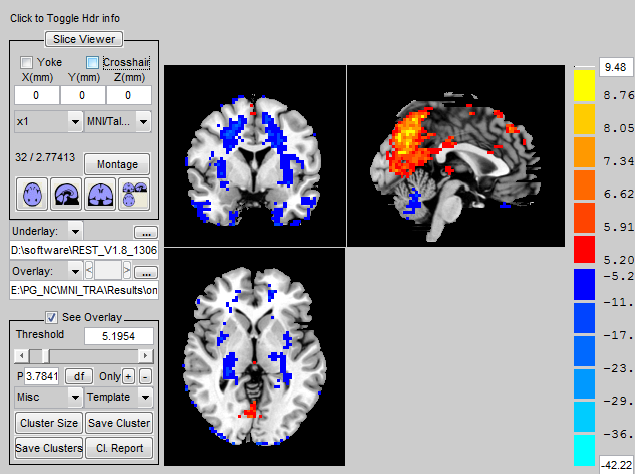
在SPM8可以成功進行到GROUP ICA 20個成分的結果
使用DPARSF過濾CSF得到的NIFTI檔 在跑GIFT的時候 14個人無法同時進行GIFT分析
會分成特定10個一起可以跑 另4個一起跑也可以 但都只能跑10個成分的結果 讓14個合併就不行
不確定是甚麼原因
錯誤訊息為
Error using load
Unable to read file 'D:\TEST_pca_r2-1.mat': no such file or directory.
而且Display幾乎沒有紅亮點 這樣是甚麼意思呢?
試了好多天 希望能為我解答 感激不盡
Hi, I am trying to set up DPARSFA to begin preprocessing my data. But when I click "Run," an error message tells me:
"Too many .nii.gz files in each subject's directory, should keep one 4D .nii.gz file."
Dear all,
I run DPARSFA starting with nifti files and so far it went more or less okay, but I get error messages for the new segment + DARTEL step:
Error using cfg_util (line 835)
Job execution failed. The full log of this run can be found in MATLAB command window, starting with
请教老师,我启动rest时总是出现下面警告,是怎么回事?
老师们好,我有几个问题想请教大家。
1、在DPARSF跑数据中,detrend这一步出错,Warning: Invalid escape sequence appears in format string. See help sprintf for valid escape sequences.
我们对病人组在治疗前后均扫描了静息态数据,也采集了健康对照组的静息态数据,在对ALFF用REST统计上犯了难,目前的试了一下,我的步骤和问题如下:
1.病人前后和对照共三组数据做ANOVA,得到了一个脑区,再在这个脑区内用REST做两两比较(该脑区为mask),发现病人治疗前该脑区内大部分voxel比对照高(REST双样本T检验),而治疗后该脑区大部分voxel比治疗前更高(用了REST的配对T检验),但是治疗后的影像与对照比却没有得到阳性发现(REST双样本T检验,跟前面是同样的多重比较矫正水平),对于这一点我就觉得纳闷,为什么治疗前比对照高,治疗后更高,治疗后比对照反而没有影像学的阳性发现?
2、我也知道不是独立样本不适合做配对T检验和ANOVA,但是有什么更合适的办法吗?或者现在我的方法有没有可以被接受的可能?
各位老师,同学好。
我现在在做GCA,用去除协变量的数据和用filtered的数据(不去除协变量)分别作了,结果不同,想知道应该用那个数据做呢,哪个更可靠呢。
谢谢。