Gretna操作
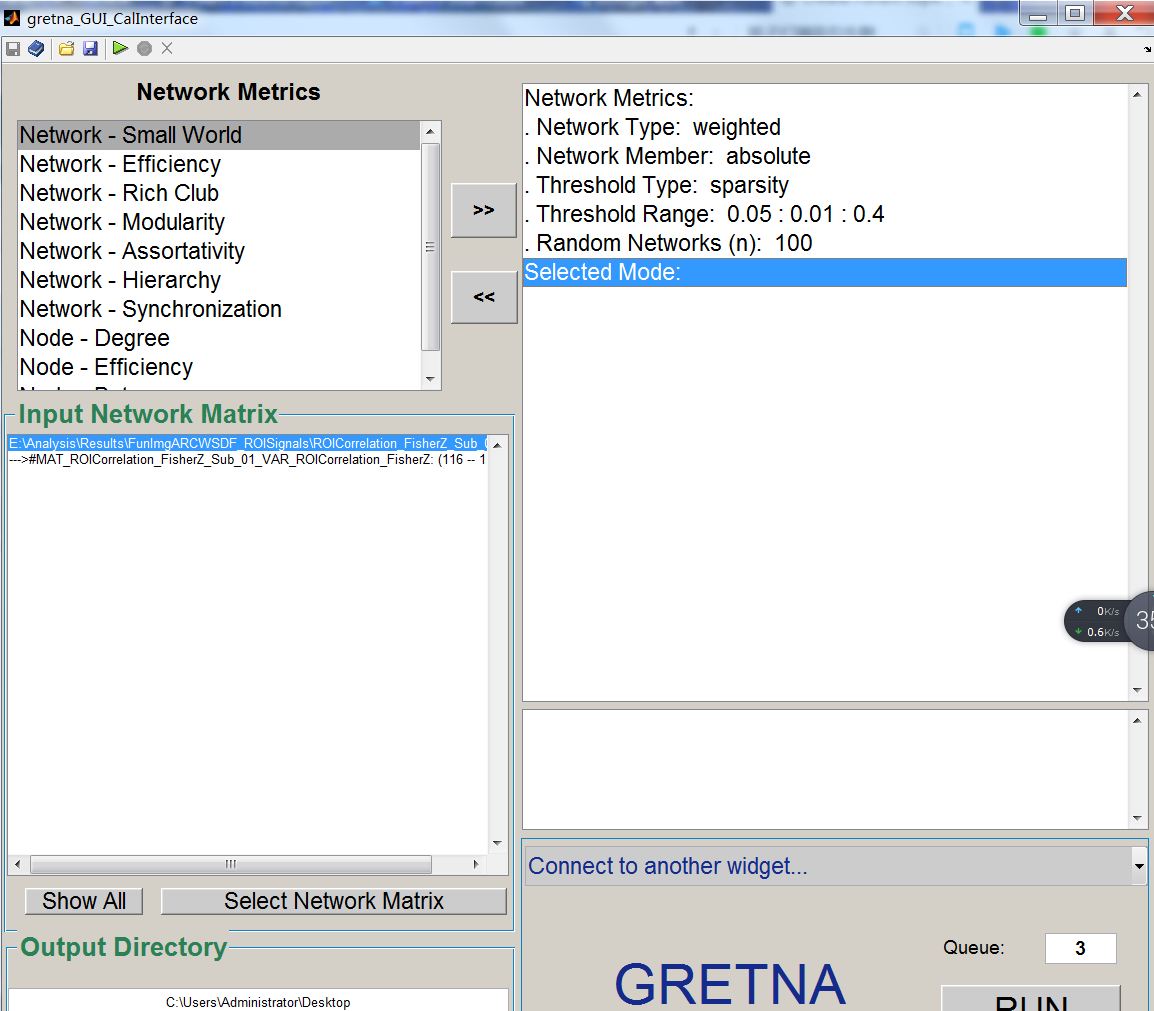
请问已经基于DPARSF得到了90*90的功能连接矩阵,使用Gretna软件计算其小世界属性描述参数时,该如何正确设置呢?
- Read more about Gretna操作
- 3 comments
- Log in or register to post comments
- 15830 reads
请问已经基于DPARSF得到了90*90的功能连接矩阵,使用Gretna软件计算其小世界属性描述参数时,该如何正确设置呢?

各位专家,大家好
我在做fMRI相关分析的时候,有文章提到说,计算两个时间序列相关性的时候,需要考虑到时间序列内部自相关(autocorrelation的影响
文章内容如下
"Because individual time points in the BOLD signal are not statistically independent, the degrees of freedom must be corrected according to Bartlett’s theory"(Michael D. Fox,PNAS,2005)
请问各位老师如果我想做3dClustSim校正应该怎么做呢?我没有AFNI这个软件,用dpabi、rest或者spm可以做吗?我看一些资料说3dClustSim比AlphaSim校正更好,不知道这两者的区别是什么呢?谢谢老师。
Dear wangyijun,
Your email you provided does not exist. Please check.
各位老师好:
我在用DPARSF软件进行静息态的fMRI数据处理,在按照严超赣老师的教程处理后,进行两样本T检验计算ReHo值的差异,用Viewer观察结果图发现: AlphaSim校正后,在非脑组织区域也存在着显著差异结果,比如蔓延到脑壳或者脑膜上,可以在我的附加图中看到,我同时也附加了HC组ReHo单样本T检验的图。
我不明白出现这种情形的原因是什么? 因为我观察在文献中都没有出现过这非脑组织表现出显著差异的情形,请问这样是正常的吗? 还是我的配准过程出现问题? 我在PicturesForChkNormalization文件夹中检查的结果有几个配的不太好的我都已经删除了被试。附件Sub_001的结果是属于绝大部分被试的配准效果,Sub_005这种配准不好的我都删除了。
所以我的疑问就是在非脑组织中出现显著差异的结果是正常的吗? 如果不正确我应该如何检查并解决问题呢?
学生刚刚接触静息态数据分析,还望各位老师能够帮助解答疑惑~
非常感谢!
Dear all:
I am trying to run ReHo analysis on my desktop computer. But I encounter error message (please see the attachment). Thanks for help.
Best Regards
Jay Chen
老师,您好!我最近在做分频段的ALFF和fALFF分析,在计算fALFF时,我对一些参数的设置不确定。
比如我想做0.01-0.027频段的fALFF分析,我在Input Parameters里输入的是不是经过0.01-0.027Hz滤波后的文件(像做ALFF一样)?还是做完去线性漂移(detrend)后的文件?
注:Option:Ideal Band Pass Filter 这一栏之所以没设,是因为我已经做完了0.01—0.027 Hz的滤波。

还有Band Hint 这一栏是不是也要设置为0.01—0.027(如下图)?其他参数设置有什么要注意的吗?

想提高双样本T检验的阳性率,之前的单样本T检验使用什么校正方法合适呢?是采用较为严格的FDR(默认全脑mask)还是采用简单的P=0.05并按照AlphaSim定义体素?
老师,您好!最近在处理一批数据,因为看了2016年的最近的静息态数据处理的视频,尝试做了一下预处理以及功能连接数据的分析,遇到了一些问题: