how to select ROI?
SORRY!I'm a newer!why and how to select ROI?
- Read more about how to select ROI?
- Log in or register to post comments
- 5355 reads
SORRY!I'm a newer!why and how to select ROI?
老师您好,我在使用slice viewer时遇到这样的情况:
按照教程的提示,我用slice viewer打开一幅ALFF双样本t检验的图,并进行了多重比较校正的相关设置。但是在看结果报表,也就是点击CL REPORT这个按钮后,不能如教程所述那样显示出相应的结果,在MATLAB中提示:
Number of clusters found: 9
??? Cell contents reference from a non-cell array object.
Error in ==> rest_sliceviewer>cuixuFindStructure at 4300
graylevel = DB{jj}.mnilist(index(ii, 1), index(ii, 2),index(ii, 3));
Error in ==> rest_sliceviewer>rest_report at 4146
[a, b] = cuixuFindStructure(tmpmni, x.DB);
Hi,
I am planning a study in which I want to measure two periods of resting state for each participants and want to compare them by means of paired-t-tests.
There are two ways of doing this: (1) stopping the scanner after the first resting period and restarting it for the second or (2) not stopping and restarting the scanner.
In task related fMRI one would not want to compare conditions that come from different scanning runs. Does the same apply for resting state? If so I assume that option (2) is better.
What would be your suggestion?
在使用Utilities中REST power spectrum时,顺利得到了某一个被试的频谱图。
但是,我想得到具体的频谱序列的值,不知道该怎么办?我查看了源代码,找到了‘theFreqSeries’这个变量,好像就是我需要的,计划增加几句代码将其输出到一个txt文件中。但感觉有点笨,不知道有没有更好的办法?或者REST软件本来已经有了,只是我没有找到?
另外,好像现在只能一个人一个人地计算频谱,我想给出两组被试各自的平均频谱,是否只能先一个一个地先算好了再计算平均值?
非常感谢各位老师在百忙中给予解答。
抱歉有两个不是问题的问题请教:
1. AFNI里AlphaSim还有关于user specified activate region情况,请问REST有考虑将来的版本升级呢?
另外请教,相应参数Zsep (separation between the mean of the noise distribution and the mean of the signal distribution)如何设定呢?
2. REST里AlphaSim关于xthr (xthr = sd*zthr + mean)计算,sd和mean都是每迭代一次,要累积前面迭代的值,我不是很明白。即如下:
count=count+nxyz; 为什么不每次迭代count都赋值为nxyz呢?
敬爱的专家:
你们好,我在做癫痫病人和对照组中分别得到了组内的单样本T检验,现在我想观察组间的T检验情况,但是我想希望单单比较癫痫组和对照组的正性连接,再比较负性连接,因为我在直接将癫痫组和对照组进行双样本T检验,如你们的数据处理视频中所说的做,我发现,有些激活的区域,两个组单样本中其实分别是正负连接,这样可能会导致统计上的放大显著差异,所以我想单单了解正性连接和负性连接的情况。但是我不知道应该怎么样通过rest实现。
希望你们看到后,能白忙中回复下,谢谢!
各位老师好,因为我是刚学,人也比较笨,所以问题很多,有些问题可能很傻,麻烦大家耐心指教啊,十分感谢!
1、因为我想做大脑的小世界性质分析,所以使用Dparsf得到了AAL90个区域的时间序列,但我想要的是90个区域的功能连接图和z-map,要怎么办?是不是要分别制作那90个ROI的mask?有没有简便的方法?
2、在制作AAL的一个ROI的mask的时候,Cluster Size是有什么用的,要怎么设置?我看了Course_Data_Process_of_Rest_fMRI_Part2_Chinese_100820这个视频但还是不明白,能否讲解一下?
3、后来我想反正我只是检验小世界性,只需要z值就行了,于是用corrcoef函数通过前面得到的AALTC求出90个区域的R值,再转换成z值,然后进行后面的计算,这种做法有没有什么不妥啊?
4、检验小世界性时,需要产生100个相应的随机网络和规则网络,是针对每个病人都要产生这100个随机网络么?我看文献上说按照马尔可夫链规则产生,那个规则具体是怎样的?就算这个规则能保证随机网络的平均度和边数与病人的网络相同,怎么能保证度分布还是相同的呢?老师那里有没有检验小世界性的做好的软件,是否是开放的?
有个小问题,我一直想不明白,请大家帮忙。
在一幅fMRI图像中,BOLD信号具体数值是多少?怎么样才能看到这个数据?
举个例子,有篇文章得到了下面的统计图,我想知道BOLD信号的数值是怎么得到的,用SPM能实现吗?
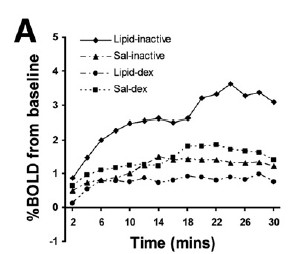
有个小问题,我一直想不明白,请大家帮忙。
在一幅fMRI图像中,BOLD信号具体数值是多少?怎么样才能看到这个数据?
举个例子,有篇文章得到了下面的统计图,我想知道BOLD信号的数值是怎么得到的,用SPM能实现吗?

Hi everybody,
I'm currently processing a series of rest fmri data which need to be manually normalized.
The procedure briefly goes like this:
-- dparsf completed the pre-normalization steps from 'dicom->nifti' to 'realign'
-- toggle spm, 'coregister: estimate',
-- switch AFNI,
'3dWarp -deoblique' and '3drefit -markers ' to generate sturctural .HEAD/BRIK pair