help 3: Rest Dicom Sorter
坛主,
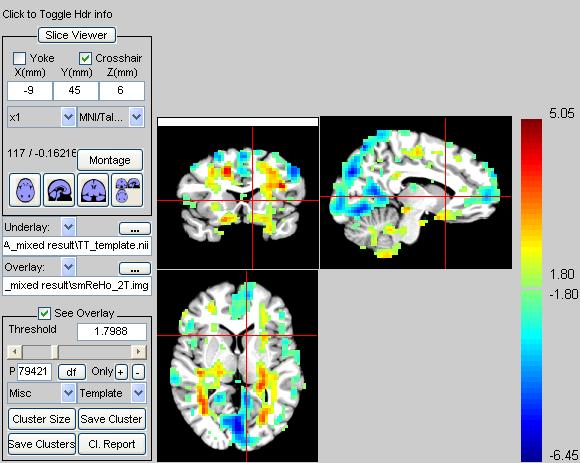
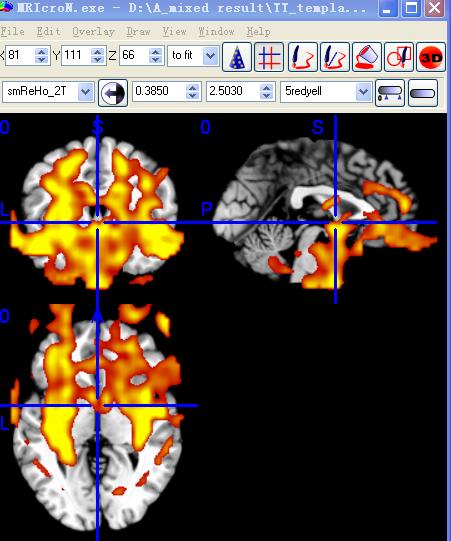
这批数据比较早,2005年GE机器,TR 1s, 16sl, 200TR.
使用Sorter时候报错:
??? Error using ==> mkdir
系统找不到指定的文件。
Error in ==> rest_DicomSorter at 38
mkdir(dirname);
- Read more about help 3: Rest Dicom Sorter
- 1 comment
- Log in or register to post comments
- 5870 reads